7月 22日,集团牛昱宇教授/季维智院士课题组在《Science Advances》(5年IF=15.388)杂志在线发表了题为“Cloning and base-editing of GFP transgenic rhesus monkey and off-target analysis”(GFP转基因猕猴的克隆、单碱基编辑与脱靶分析)的研究论文。该工作基于体细胞核移植(SCNT)技术,建立了一种适用于灵长类动物的单碱基编辑器脱靶检测方法OA-SCNT,并利用该方法对腺嘌呤单碱基编辑器ABEmax在猕猴胚胎DNA、RNA水平的脱靶效应进行了系统评估。
牛昱宇教授、季维智院士为本文通讯作者,学院在站博士后康宇、王芳,代绍兴副教授,博士研究生曾玉强为文章并列第一作者。

单碱基编辑器(base editors)是治疗由点突变产生的遗传性疾病最有效和精确的方法,由于能够在不依赖DNA双链断裂(DSB)的情况下实现对单个碱基的定向修改,单碱基编辑器能够最大限度的减少基因编辑中不良产物的产生。然而单碱基编辑器依然存在一定的脱靶效应,并由此带来潜在的安全性问题。由于个体单核苷酸多态性(SNP)的存在,单碱基编辑器的脱靶效应评估依赖于高灵敏度的检测技术,同时需要有严格的对照样本,才能够从全基因组测序数据中识别出非 sgRNA依赖的脱靶突变。2019年,《Science》杂志上发表的两篇文章分别用不同的方法在水稻和小鼠上证明了单碱基编辑系统存在严重的脱靶效应。由于灵长类动物在遗传和生理上与人类高度相似,在应用于临床之前,基因编辑工具在灵长类动物个体水平的效率和安全性评估显得尤为重要。
在本项工作中,研究者以一只表达绿色荧光蛋白(GFP)的成年转基因猕猴的体细胞核作为供体构建克隆胚胎。在胚胎早期阶段利用单碱基编辑沉默 GFP表达,并通过胚胎移植得到克隆猴。由于细胞核来自同一只动物,编辑组和对照组样本的基因背景完全一致,通过比对两者之间的差异便可鉴定出由基因编辑工具造成的突变。
研究者收集了包含克隆胚胎、克隆猴成体组织在内的样本进行全基因组和转录组测序,进而完成DNA和RNA水平的脱靶分析。在DNA水平,无论是 SNV总数还是特异的 A > G和T > C突变数量,编辑组与对照组均没有显著差异,说明 ABEmax 系统并没有引起明显的 DNA脱靶编辑,这一点与小鼠胚胎的结果类似。然而 RNA脱靶分析结果则显示,在编辑组的猕猴胚胎中出现了显著增加的 SNVs,且绝大多数为 A > G和T > C突变,表明 ABEmax会引起大规模的 RNA脱靶编辑。在基因表达总体水平相当的情况下,脱靶突变更多发生在表达量相对较高的基因,并且随机分布在不同的转录本中。此外,分析数据表明,98%以上的脱靶突变发生在蛋白编码序列,仅 1.9%为长链非编码 RNA(lncRNAs)。研究者进一步评估了 RNA脱靶突变的基因与胚胎早期发育关键基因的关系,发现每个胚胎中平均有392个脱靶编辑突变发生在与胚胎早期发育相关的基因,但对基因表达量没有影响。此外,每个编辑组胚胎样本中平均有 109个脱靶编辑位点与致癌基因或抑癌基因相关,同样,这些基因的表达量与对照组相比没有显著变化,但其蛋白编码序列有可能被改变。
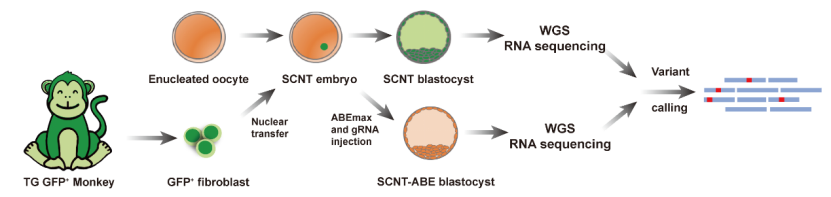

(上图:OA-SCNT方法示意图;下图:供体猴及克隆猴照片)
综上,该研究首次成功实现了成年转基因猕猴的克隆和单碱基编辑,并以此为基础建立了灵长类动物单碱基编辑脱靶分析方法,首次实现了灵长类个体水平的单碱基编辑脱靶效应评估,将为精准基因编辑工具的开发和优化提供重要数据。同时,该文章也是牛昱宇/季维智研究团队继3 月4 日之后,本年度第二篇发表在《Science Advances》上的研究论文,两篇文章均基于灵长类基因编辑技术展开,为利用灵长类动物开展的大脑发育、疾病发生机制和治疗研究提供了重要参考。
原文链接:
https://science.org/doi/10.1126/sciadv.abo3123
